Artikkel: Standard ingeniørprinsipper som prediktivt rammeverk for biologi
Av Emily Reeves; 3. januar 2024. Overrsatt herfra
 I 2017 publiserte professor i Ingeniørfag Gregory T. Reeves og ingeniør Curtis E. Hrischuk en åpen tilggjengelig artikkel i Journal of Bioinformatics, Computational and Systems Biology med tittelen (oversatt) "Cellen legemliggjør standard ingeniørprinsipper." De forklarte hvordan cellen oppfyller forskjellige sett av Standard IngeniørPrinsipper (SIP-er). Denne artikkelen bygger på Reeves og Hrischuks tidligere publikasjon som undersøkte ingeniørmodeller for systembiologi. Nok en gang hevder disse forfatterne at ingeniørkonsepter kan brukes som et prediktivt og fruktbart rammeverk for biologi.
I 2017 publiserte professor i Ingeniørfag Gregory T. Reeves og ingeniør Curtis E. Hrischuk en åpen tilggjengelig artikkel i Journal of Bioinformatics, Computational and Systems Biology med tittelen (oversatt) "Cellen legemliggjør standard ingeniørprinsipper." De forklarte hvordan cellen oppfyller forskjellige sett av Standard IngeniørPrinsipper (SIP-er). Denne artikkelen bygger på Reeves og Hrischuks tidligere publikasjon som undersøkte ingeniørmodeller for systembiologi. Nok en gang hevder disse forfatterne at ingeniørkonsepter kan brukes som et prediktivt og fruktbart rammeverk for biologi.
Menneskelig utforming og bygning har resultert i lister over standard ingeniørprinsipper som må følges for å produsere effektive, robuste systemer. Disse prinsippene er blitt foredlet gjennom utallige ingeniørprosjekter, og Reeves og Hrischuk demonstrerer at de samme SIP-ene brukes i biologi. De er derfor nyttige for biologer som et forventnings-rammeverk for forventninger om cellulære Systemer:
"Tilstedeværelsen av ingeniørprinsipper i cellen innebærer at SIP-er kan brukes som utgangspunkt for å formulere hypoteser om hvordan en celle opererer og oppfører seg. Med andre ord, vi bør pragmatisk nærme oss cellen som et konstruert system og bruke det synspunktet for å forutsi (antyde) den forventede oppførselen til biologiske systemer. Vi kaller denne tilnærmingen IngeniørPrinsippets Forventning (IPF).
Bilde 1. Cellestruktur (nettverk)
Flere kategorier av SIP-er
I artikkelen blir flere kategorier av SIP-er undersøkt: Generelle IngeniørPrinsipper (GIPer), maskinvare/programvare-KoDesignPrinsipper (KDePer) og RobotTeknikk-Prinsipper (RTPer). For hver av disse kategoriene gir forfatterne spesifikke eksempler på hvordan cellen samsvarer med settet med SIP-er. Forfatterne utvikler også en ikke-uttømmende liste over SIP-er for Kjemisk Prosess Kontroll Ingeniør-Prinsipper (KPKIPer), siden en liste ikke var tilgjengelig.
 Sammenligningen mellom cellulære systemer og konstruerte systemer har sterke implikasjoner for intelligent design. Realiteten som celler overholder de samme ingeniørprinsippene som er oppdaget i menneskelig design, er svært betydelig. Dette funnet er mye bedre varslet i hypotesen om at biologiske systemer er intelligent designet, enn den alternative teorien om en blind neo-darwinistisk prosess, som gir opphav til levende systemer.
Sammenligningen mellom cellulære systemer og konstruerte systemer har sterke implikasjoner for intelligent design. Realiteten som celler overholder de samme ingeniørprinsippene som er oppdaget i menneskelig design, er svært betydelig. Dette funnet er mye bedre varslet i hypotesen om at biologiske systemer er intelligent designet, enn den alternative teorien om en blind neo-darwinistisk prosess, som gir opphav til levende systemer.
For kategorien generelle ingeniørforventninger går forfatterne gjennom tre prinsipper i hovedteksten. GIP1 uttaler at "utvikling av konstruerte objekter følger en plan i samsvar med kvantitative krav." Forfatterne påpeker at utviklingen av molekylære maskiner krever nøye orkestrering, inkludert, men ikke begrenset til, beslutningstaking, genuttrykk, proteinsyntese, post-translasjonell modifisering, montering av multikomponent-komplekser og livssyklus-prosesser som celledeling -lenke. Dermed legemliggjør celler GIP1. GIP2 uttaler at "krav rangeres i henhold til kostnadseffektivitet, og utviklingsplan, som har en trinnvis struktur, understreker de høyere rangerte kravene." Dette prinsippet beskriver hierarki, som er resultatet av et Top-Down design der komponenter er konstruert, og ressurser brukt i samsvar med høyere systemmål. For å gi et biologisk eksempel, trekker forfatterne frem prioritering av ATP i cellen. GIP3 sier at "standarder brukes hvor tilgjengelig, og aktuelle ved alle utganger fra gjeldende standarder eksplisitt er berettiget." Biologiske eksempler inkluderer hvordan alle forskjellige typer celler har bevart funksjoner som den genetiske koden, ATP, en nær universell sentral metabolisme. Aminosyrer, nukleinsyrer og noen lipider kan alle tenkes som en cellulær standard som avvik sjelden forekommer fra.
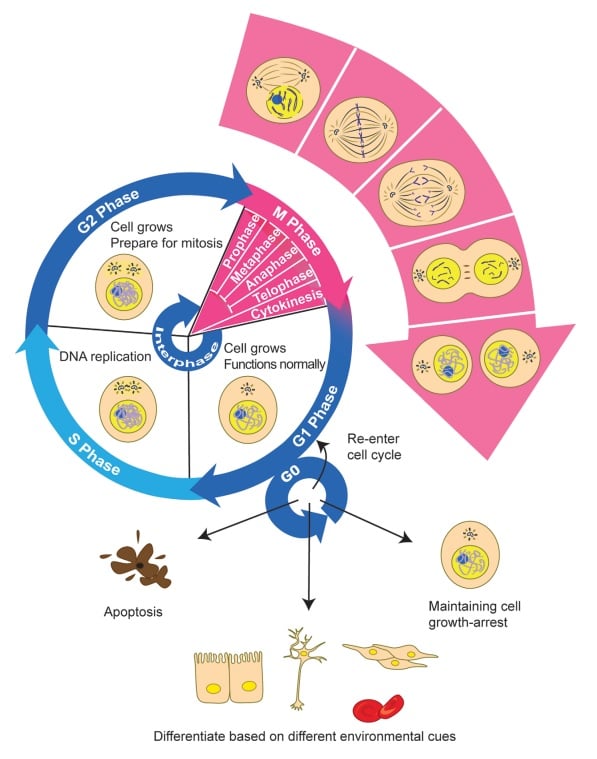
Bilde 2. Cellens livssyklus
Maskinvare/programvare-sammensetningsprinsipper
Deretter diskuterer forfatterne Maskin-Programvare-CoDesign-Prinsipper som starter med CDeP1. Dette er prinsippet om å "dele opp funksjonen som skal implementeres, i små interaksjonelle stykker." I cellen kan cellulære regulatoriske nettverk avsøres i autonomt virkende moduler som samarbeider, for å utføre en funksjon. Til og med det grunnleggende om cellulær fysiologi, der unike makromolekylære strukturer som kromosomer, membraner og ribosomer eksisterer, innebærer partisjonering av funksjonen i små interagerende stykker. Dermed florerer celler med eksempler på autonome utøvere som utfører en spesifikk rolle, mot et større formål. CDeP2 er prinsippet om å "tildele disse partisjonene til mikroprosessorer eller andre maskinvareenheter, der funksjonen kan implementeres direkte i maskinvare eller i programvare som kjører på en mikroprosessor." Dette prinsippet ligger til grunn for fordelen med å ha en egen prosessor for hver funksjon. I datasystemer utelukker produksjonsbegrensninger dette fra å være mulig, men forfatterne påpeker at cellen er i stand til å realisere idealet om å ha hvert protein eller kompleks som fungerer uavhengig, som en unik enhet maskinvare.
 Reeves og Hrischuk beskriver deretter RIPer og KDePer. Mens det å gå gjennom hver av disse, er utenfor synsvidden til denne artikkelen, er takeaway at SIP-er gir logikk til å forstå biologiske systemer. Ved å gjøre seg kjent med disse prinsippene, kan biologer forbedre sine forskningsmetodologier og forbedre sin evne til å forutsi og validere sine eksperimenter.
Reeves og Hrischuk beskriver deretter RIPer og KDePer. Mens det å gå gjennom hver av disse, er utenfor synsvidden til denne artikkelen, er takeaway at SIP-er gir logikk til å forstå biologiske systemer. Ved å gjøre seg kjent med disse prinsippene, kan biologer forbedre sine forskningsmetodologier og forbedre sin evne til å forutsi og validere sine eksperimenter.
Ingeniørprinsippets forventning
Reeves og Hirschuk sier at et hvilket som helst komplekst system må knytte seg til SIPer. Hvis de ikke gjør det, er resultatet katastrofalt. Biologiske systemer, som er mer komplekse enn noe konstruert system i dag, er ikke noe unntak. Når man ser på et biologisk system, bør man forvente ingeniøregenskaper. Dette kan tenkes som ingeniørprinsippets forventning, en prediktiv modell som kan brukes når du ser på et biologisk system hvis mekanistiske detaljer ikke blir forstått. Reeves og Hrischuk hevder at det er avgjørende å anvende ingeniørprinsipper for å forstå og analysere biologiske systemer. Ved å gjøre det, kan forskere få innsikt i de underliggende mekanismene og forutsi atferden til disse systemene. I tillegg kan det å konsumere ingeniørprinsipper hjelpe til med å utforme effektive intervensjoner eller terapier for komplekse biologiske problemer.
Bilde 3. Felles begrepsbruk i systembiologi og ingeniørfag
 Emily Reeves
Emily Reeves
Emily Reeves er en biokjemiker, metabolsk ernæringsfysiolog og aspirerende systembiolog. Hennes doktorgradsstudier ble fullført ved Texas A&M University i biokjemi og biofysikk. Emily er for tiden en aktiv kliniker for metabolsk ernæring og ernæringsmessig genomikk ved Nutriplexity. Hun liker å identifisere og designe ernæringsintervensjon for subtile medfødte metabolismefeil. Hun jobber også med stipendiater fra Discovery Institute og det større vitenskapelige samfunnet for å fremme integrering av ingeniørfag og biologi. Hun tilbringer helgene på oppdagingsferd med mannen sin, brygge Kombucha og løpe i nærheten av Puget Sound.
Musikk til Guds ære: Forever.
Overrsettelse via google oversetter og bilder ved Asbjørn E. Lund
 I 2017 publiserte professor i Ingeniørfag Gregory T. Reeves og ingeniør Curtis E. Hrischuk en åpen tilggjengelig artikkel i Journal of Bioinformatics, Computational and Systems Biology med tittelen (oversatt) "Cellen legemliggjør standard ingeniørprinsipper." De forklarte hvordan cellen oppfyller forskjellige sett av Standard IngeniørPrinsipper (SIP-er). Denne artikkelen bygger på Reeves og Hrischuks tidligere publikasjon som undersøkte ingeniørmodeller for systembiologi. Nok en gang hevder disse forfatterne at ingeniørkonsepter kan brukes som et prediktivt og fruktbart rammeverk for biologi.
I 2017 publiserte professor i Ingeniørfag Gregory T. Reeves og ingeniør Curtis E. Hrischuk en åpen tilggjengelig artikkel i Journal of Bioinformatics, Computational and Systems Biology med tittelen (oversatt) "Cellen legemliggjør standard ingeniørprinsipper." De forklarte hvordan cellen oppfyller forskjellige sett av Standard IngeniørPrinsipper (SIP-er). Denne artikkelen bygger på Reeves og Hrischuks tidligere publikasjon som undersøkte ingeniørmodeller for systembiologi. Nok en gang hevder disse forfatterne at ingeniørkonsepter kan brukes som et prediktivt og fruktbart rammeverk for biologi. Sammenligningen mellom cellulære systemer og konstruerte systemer har sterke implikasjoner for intelligent design. Realiteten som celler overholder de samme ingeniørprinsippene som er oppdaget i menneskelig design, er svært betydelig. Dette funnet er mye bedre varslet i hypotesen om at biologiske systemer er intelligent designet, enn den alternative teorien om en blind neo-darwinistisk prosess, som gir opphav til levende systemer.
Sammenligningen mellom cellulære systemer og konstruerte systemer har sterke implikasjoner for intelligent design. Realiteten som celler overholder de samme ingeniørprinsippene som er oppdaget i menneskelig design, er svært betydelig. Dette funnet er mye bedre varslet i hypotesen om at biologiske systemer er intelligent designet, enn den alternative teorien om en blind neo-darwinistisk prosess, som gir opphav til levende systemer. Reeves og Hrischuk beskriver deretter RIPer og KDePer. Mens det å gå gjennom hver av disse, er utenfor synsvidden til denne artikkelen, er takeaway at SIP-er gir logikk til å forstå biologiske systemer. Ved å gjøre seg kjent med disse prinsippene, kan biologer forbedre sine forskningsmetodologier og forbedre sin evne til å forutsi og validere sine eksperimenter.
Reeves og Hrischuk beskriver deretter RIPer og KDePer. Mens det å gå gjennom hver av disse, er utenfor synsvidden til denne artikkelen, er takeaway at SIP-er gir logikk til å forstå biologiske systemer. Ved å gjøre seg kjent med disse prinsippene, kan biologer forbedre sine forskningsmetodologier og forbedre sin evne til å forutsi og validere sine eksperimenter. Emily Reeves
Emily Reeves